
环状RNA在食道鳞状细胞癌(ESCC)等许多癌症中发挥着重要作用,但大多数环状RNA的确切功能尚不清楚。2023年1月12日,云序客户山东大学齐鲁医院孙振国团队在Cell Death Discov(IF=7.109)上发表文章“Circular RNA circTRPS1-2 inhibits the proliferation and migration of esophageal squamous cell carcinoma by reducing the production of ribosomes”通过对ESCC组织和邻近正常组织的研究,揭示了circTRPS1-2可以通过减少核糖体的产生来抑制ESCC的增殖和迁移,从而确立了其在ESCC治疗和预后预测中的潜在作用。云序生物有幸参与了此项研究中的全转录组测序高通量测序技术服务。

发表期刊:Cell Death Discov
影响因子:7.109
发表时间:2023年1月12日
研究方法:全转录组测序、环状RNA RT-PCR、RIP-PCR、RNA pull down等
文章链接:Circular RNA circTRPS1-2 inhibits the proliferation and migration of esophageal squamous cell carcinoma by reducing the production of ribosomes
云序提供服务:

技术路线图

研究内容
1.circTRPS1-2在ESCC组织中的表达
对3对ESCC和邻近的正常组织进行全转录组高通量测序,测序所得的circRNA结果发现2685个环状RNA在癌症中上调,4881个环状RNA在癌症下调(图1a,b)。进一步设定差异倍数∣fold change ∣≥2和P < 0.05进行筛选,最终确定了5个上调的环状RNA和41个下调的环状RNA(图1c)。此后,文章用30对独立的ESCC和邻近的正常组织验证了5个上调的环状RNA和以下10个下调最多的环状RNA,hsa_circ_0026782,hsa_circ_0085362(circTRPS 1-2)和hsa_circ_0078299等(图1d)。
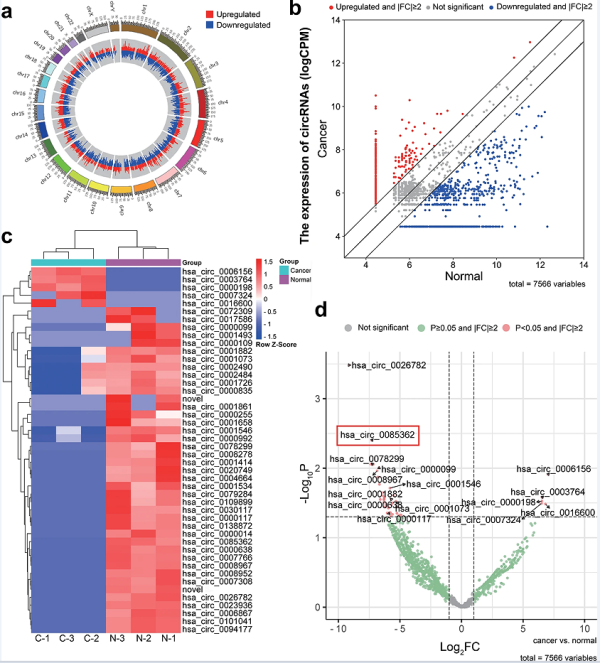
图1 ESCC组织的高通量测序鉴定circTRPS1-2
2.ESCC细胞系中circTRPS1-2的下调和亚细胞定位
根据UCSC基因组浏览器(GRCh37/hg19)比对结果分析,CircTRPS1-2位于染色体8:116599227-116635985上(图2a),长2821 bp,由TRPS1基因的第2外显子与第5外显子反向剪接产生(图2b)。此外,circTRPS1-2在K150和E109细胞系中均表达下调(图2c)。随后文章作者通过对qRT-PCR产物的Sanger测序,验证了反向剪切(图2d)。并且分歧引物能够从cDNA中扩增circTRPS1-2,但不能从基因组DNA中扩增,而聚合引物能够从cDNA和基因组DNA中扩增线性TRPS1(图2e)。这些结果支持了circTRPS1-2是通过反向剪接而不是反式剪接或基因组重排而产生的观点。circTRPS1-2的环状结构通过其对RNase R消化的耐受性得到证实(图2f)。根据circTRPS1-2的FISH(图2g)和核-细胞质分离(图2h),发现主要定位在细胞质和细胞核中。
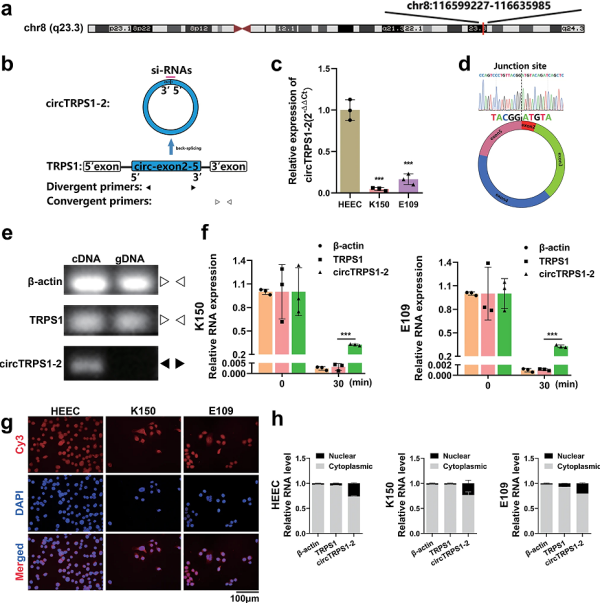
图2 circTRPS1-2在ESCC组织中的特征和亚细胞定位
3.ESCC肿瘤中circTRPS1-2的下调及其与不良预后的关系
在30例ESCC和对应的邻近患者的正常组织中,文章发现circTRPS1-2在肿瘤中显著下调(图3a)。BaseScope实验也得到了类似的结果,环状RNA主要定位于细胞质和细胞核(图3b,c)。30例患者中,circTRPS1-2表达低于中位水平的患者的3年总生存期明显低于表达高于中位水平的患者(P = 0.025;图3d)。这些结果表明,circTRPS1-2可能作为一种抗ESCC的肿瘤抑制因子,其低表达预示着预后不良。
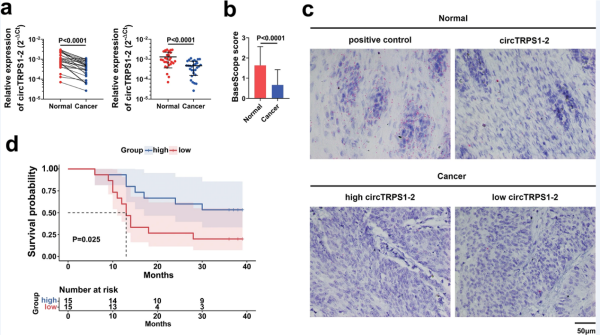
图3 circTRPS1-2在ESCC组织中的下调及其表达与预后呈正相关
4.CircTRPS1-2抑制ESCC细胞系的增殖和迁移
为了研究circTRPS1-2在ESCC细胞中的生物学功能,文章用两个ESCC细胞系分别转染过表达circTRPS1-2的质粒和用siRNAs敲除(图4a)。这些转染对线性TRPS1的表达没有显著影响(图4b)。根据集落形成(图4c,d)、EdU掺入(图4e,f)和细胞计数试剂盒Kit-8(图4g)的分析,过表达circTRPS1-2显著降低了ESCC细胞的活力和增殖。根据伤口愈合试验(图4h,i)和跨孔迁移(图4j、k),也显著降低了ESCC细胞的迁移能力。而敲除circTRPS1-2的效果则相反。
图片
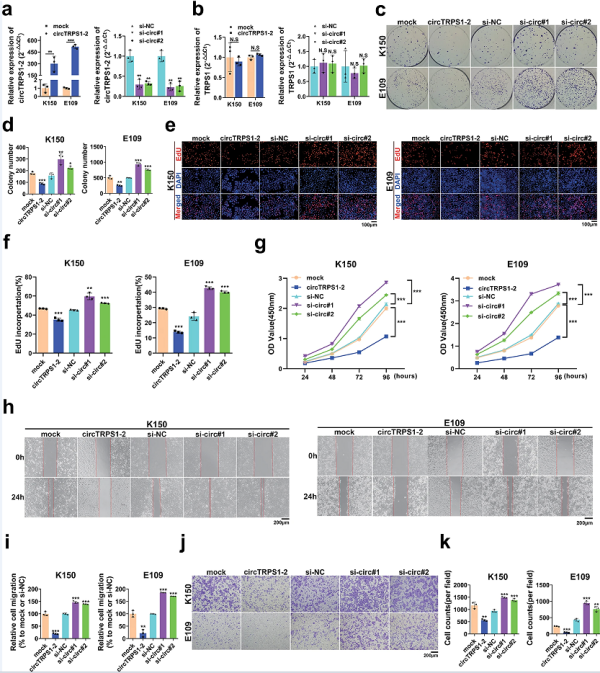
图4 通过circTRPS1-2抑制ESCC细胞的增殖和迁移
5.CircTRPS1-2在体内抑制肿瘤的发生和转移
在皮下肿瘤实验中(图5a),circTRPS1-2的过表达导致肿瘤体积(图5b)和重量(图5c)明显变小。敲除circTRPS1-2则产生了相反的作用。在肿瘤转移实验中,circTRPS1-2的敲除显示出明显更多的浅表转移(图5d)、肺转移(图5e、f)和表面和深部的整体转移(图5g)。过表达circTRPS1-2可减少肺转移(图5f)和总转移(图5g)。
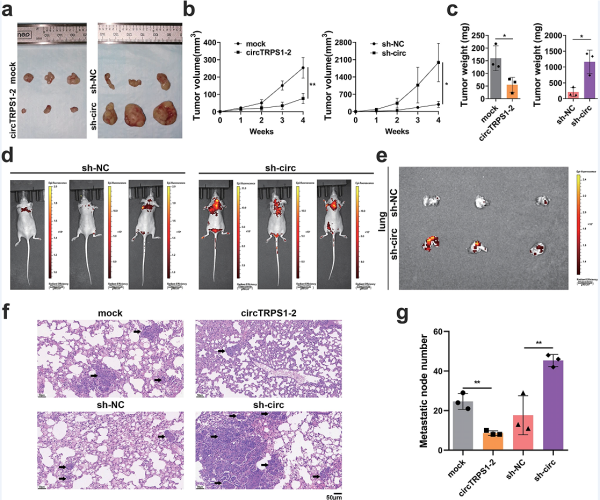
图5 CircTRPS1-2在体内抑制ESCC的肿瘤发生和转移
6.CircTRPS1-2可能调节核糖体的生物发生
为了阐明circTRPS1-2可能在ESCC中作为肿瘤抑制因子的途径,在排除了26个正义和反义RNA探针拉下的蛋白质后,文章继续分析了15个与正义探针特异性结合的蛋白质(图6a)。GO分析和KEGG分析结果表明,这15个蛋白与核仁和细胞质中的核糖体和核糖核RNA加工密切相关(图6b,c)。所以文章推测circTRPS1- 2可能参与了核糖体的生物发生,因此文章作者随后检测了15个候选相互作用中的5个核糖体蛋白:S4X、S8、S24、L7和L11。最后通过RNA pull down下拉后的蛋白印迹实验证实了circTRPS1-2能直接与这些蛋白结合(图6d)。此外,使用RIP-PCR(图6e)以及免疫荧光染色(图6f),根据260 nm处的吸光度,发现过表达导致细胞提取物中核糖体水平降低,下调较高(图6g)。电镜观察显示,过表达导致核糖体数量和核仁减少,而敲除后的作用则相反(图6h)。
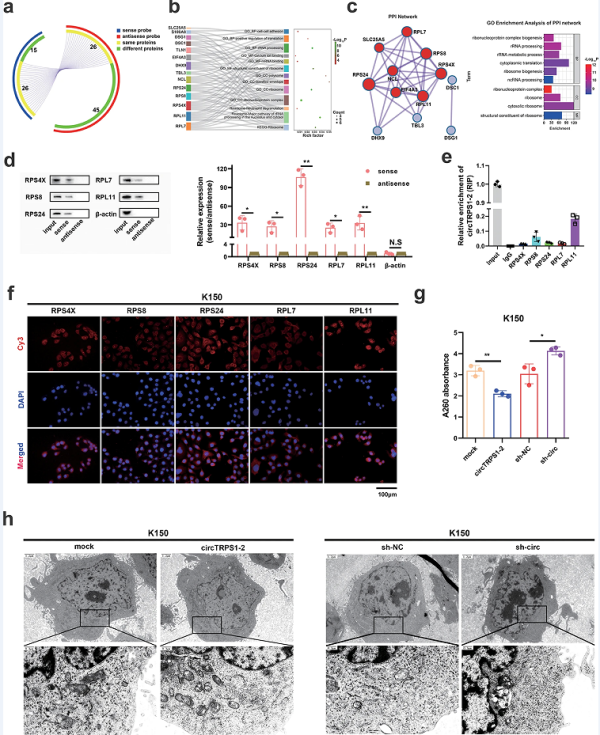
图6 circTRPS1-2与核糖体蛋白的结合及其对核糖体生物发生的影响
总 结
文章联合环状RNA测序、环状RNA qRT-PCR、RIP-PCR、RNA pull down等技术证明circTRPS1-2通过与核糖体蛋白相互作用以此调节核糖体的产生并发挥抑瘤作用,并探索circTRPS1-2作为潜在的预后生物标志物和治疗靶点在ESCC中的作用。
云序环状RNA课题技术服务四大模块
一、 环状RNA筛选:
1.1 circRNA测序
1.2 全转录组测序(同时检测circRNA、lncRNA、mRNA)
二、环状RNA验证
2.1 qRT-PCR
对选定目标RNA分子可进行qPCR检测服务,包括引物设计与表达鉴定。
2.2 Sanger测序与RNase R酶耐受实验
对选定的环状RNA分子进行反式剪切位点引物设计,扩增后Sanger测序验证环状;使用RNase R酶处理环状RNA分子,验证环状RNA分子稳定性。
三、 CircRNA功能机制研究
3.1 RIP测序/RIP-qPCR
针对目标蛋白抗体把蛋白-RNA复合物沉淀下来,并对复合物上的RNA进行测序或对目标RNA进行qRCR表达分析。
3.2 RNA pull-down+MS/WB/qPCR/RNA-Seq
设计目标RNA生物探针把相应的结合蛋白质与RNA复合物沉淀下来,蛋白质谱检测或WB检测与RNA结合的蛋白。设计目标RNA生物探针把相应的结合蛋白质与RNA复合物沉淀下来,qPCR检测或RNA-seq检测与RNA结合的RNA。
3.3 ChIP-seq/ChIP-PCR
检测转录因子或者组蛋白与下游基因的结合情况。
四、CircRNA修饰研究
4.1 m6A/m5C/ac4C/m7G/m1A等修饰
针对环状RNA各类修饰进行高通量筛选。
4.2 MeRIP-circRNA-PCR验证
对高通量测序的结果,通过qPCR进行进一步验证。
云序客户部分环状RNA相关文章

相关产品
全转录组测序(同时检测circRNA、lncRNA、mRNA)
circRNA测序
RIP测序
RNA pull down
环状RNA qRT-PCR
Sanger测序与RNase R酶耐受实验
MeRIP-circRNA-PCR验证

上海云序生物科技有限公司
地址: 松江区莘砖公路518号18号2楼
网址: http://www.cloud-seq.com.cn
电话: 021-64878766
邮箱: market@cloud-seq.com.cn
(正文已结束)
免责声明及提醒:此文内容为本网所转载企业宣传资讯,该相关信息仅为宣传及传递更多信息之目的,不代表本网站观点,文章真实性请浏览者慎重核实!任何投资加盟均有风险,提醒广大民众投资需谨慎!
| 您看到此文《用户文章|环状RNA测序助力食道鳞状细胞ai的机制研究》感受(已有 8 人表态) | ||||||||||||||
 欠扁 |  同意 |  很好 |  胡扯 |  搞笑 |  软文 |  糊涂 |  惊讶 | |||||||




